拉曼光谱解码:机器学习助力无标记SERS快速鉴定病原菌
发布时间:2025-04-07 浏览次数:94
1. 引言
快速准确的病原体检测至关重要,但传统方法存在局限性。表面增强拉曼散射(SERS)技术具有灵敏、快速、无损等优势,尤其无标记SERS无需复杂标记,可直接检测细菌的固有振动指纹。然而,SERS分析面临信号峰重叠和咖啡环效应导致信号不均匀等挑战。为解决这些问题,机器学习(ML)与数据预处理技术结合,可有效去除噪声和背景干扰,提高SERS数据分析的准确性,为病原菌的快速鉴定提供了新的途径。
本研究报道了一种结合机器学习和数据预处理的无标记疏水SERS平台,用于病原菌的快速高通量检测。首先优化了SERS信号,并比较了咖啡环效应和疏水富集效应下的SERS性能。然后,针对SERS数据开发并评估了四种机器学习分类模型:k-NN、SVM-poly、SVM-rbf和1D-CNN。其中,SVM模型被广泛应用于数据分类任务,而CNN模型则擅长学习数据的空间结构和模式。此外,k-NN模型作为一种简单的无监督学习算法也被应用于此研究。
2. 结果与讨论
使用疏水SERS平台进行细菌检测的无标签SERS分析
本研究利用疏水SERS平台进行病原菌的无标记快速检测。疏水硅片通过限制液滴扩散,使细菌和纳米颗粒集中,产生“局部浓度效应”,相比于普通硅片上的“咖啡环效应”,更有利于SERS检测。研究人员用该平台对大肠杆菌、单核增生乳杆菌、鼠伤寒沙门氏菌和金黄色葡萄球菌四种食源性致病菌进行了检测,并构建了包含800个光谱的数据集。通过数据预处理技术对数据集进行标准化,再结合机器学习分类模型,实现了对这四种细菌的准确分类。

图1. 使用疏水SERS平台进行细菌检测的无标签SERS分析
细菌和等离子体粒子混合物在正常和疏水硅衬底上的分布及PNs的表征
利用金核银壳纳米粒子(Au@AgNPs)增强SERS信号,并比较了普通硅片和疏水硅片作为基底的效果。Au@AgNPs结构均匀,在普通硅片上,由于咖啡环效应,Au@AgNPs和细菌集中在液滴边缘;而在疏水硅片上,则集中在中心区域,形成“局部浓度效应”,显著提高了细菌和Au@AgNPs的浓度。疏水处理后的硅片接触角明显增大,证实了其良好的疏水性。结果表明,疏水基底更有利于SERS检测,因为它能增加热点成分并使细菌充分暴露于热点中。

图2. 细菌和等离子体粒子混合物在正常和疏水硅衬底上的分布及PNs的表征
咖啡环效应和局部浓度效应下不同PNs配比的SERS活性优化
比较了咖啡环效应和局部浓度效应下Au@AgNPs对大肠杆菌的SERS检测效果。结果表明,疏水硅片上的局部浓度效应比普通硅片上的咖啡环效应具有更高的灵敏度。在734 cm⁻¹处,局部浓度效应的最佳Au@AgNPs浓度为2.6 μg/mL,而咖啡环效应则为21 μg/mL。此外,局部浓度效应下的SERS强度比咖啡环效应高33倍,且检测限更低。这表明疏水硅片更有利于提高SERS性能,因为它能有效提高细菌和Au@AgNPs的局部浓度。

图3. 咖啡环效应和局部浓度效应下不同PNs配比的SERS活性优化
数据预处理和数据集准备
利用机器学习模型对四种细菌的SERS数据进行分类。由于原始SERS数据存在噪声干扰,研究人员采用了基线校正、平滑和归一化等预处理步骤,增强了特征峰谱的清晰度。通过比较k-NN、SVM-poly、SVM-rbf和1D-CNN四种ML模型的分类性能,研究探索了不同细菌拉曼光谱的内在差异。此外,通过分析预处理后鸟嘌呤峰和腺嘌呤峰的相对强度,验证了在不使用ML模型的情况下,利用SERS光谱区分细菌的可行性。
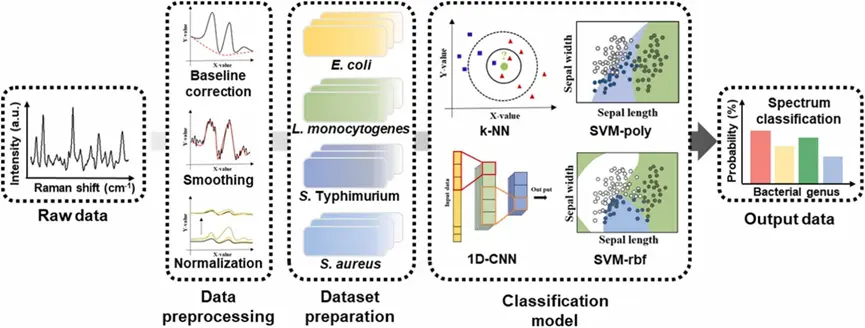
图4. 数据预处理流程图,数据集准备,机器学习分类模型。
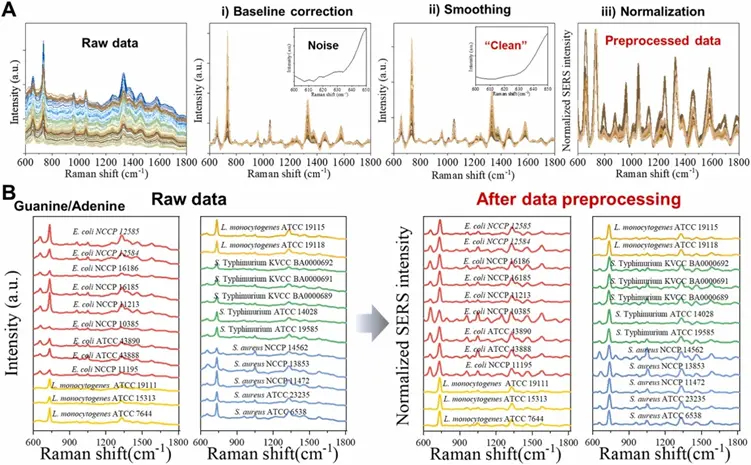
图5. 数据预处理和数据集准备
研究发现,不同细菌的鸟嘌呤/腺嘌呤峰值比存在差异,可用于区分部分细菌,但同种细菌的不同菌株之间也存在信号差异,例如金黄色葡萄球菌菌株间差异较大,难以区分。为评估疏水SERS平台的再现性,对大肠杆菌进行了40次重复测量,相对标准偏差为7.45%。此外,不同培养温度(25-42℃)下,疏水基底均能产生均匀的拉曼光谱,但42℃时腺嘌呤峰信号减弱,可能与细菌成分或代谢物变化有关。结果表明,疏水基底的局部浓度效应保证了SERS信号的稳定性和可重复性。
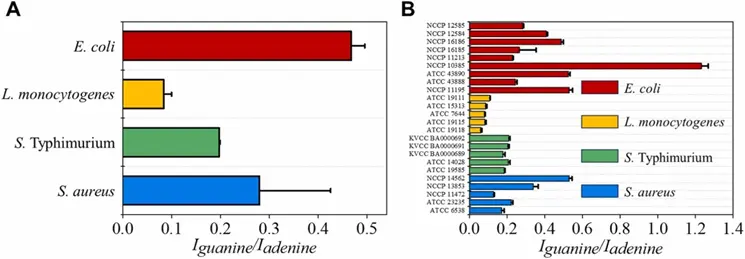
图6. (A)种和(B)菌株水平上的鸟嘌呤相对强度峰比(654 ~ 661 cm−1)和腺嘌呤相对强度峰比(734 ~ 737 cm−1)。数据为20个拉曼光谱的平均值±标准差。
使用ML模型的细菌分类结果
本研究使用k-NN、SVM-poly、SVM-rbf和1D-CNN四种机器学习模型对预处理后的SERS数据进行细菌分类。结果显示,数据预处理,尤其是基线校正、平滑和归一化,显著提高了模型分类准确率。未经预处理的原始数据中,除k-NN外,其他模型均出现错误分类。ROC曲线分析也表明,预处理后模型的AUC值显著增加。即使在咖啡环效应下,预处理后的SERS光谱准确率也能达到95%以上。研究证实,数据预处理对于基于SERS数据和机器学习的细菌分类至关重要。
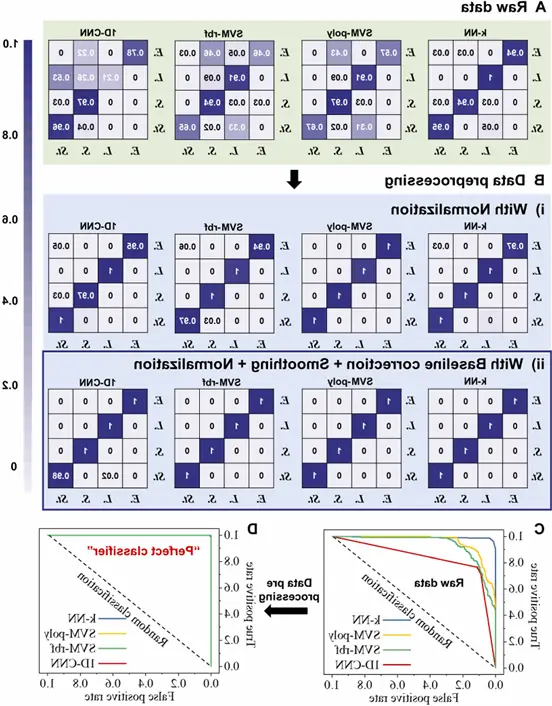
图7. (A)原始数据和(B)预处理后的数据得到k-NN、SVM-poly、SVM-rbf和1D-CNN四种分类模型的混淆矩阵。E., L., S., St.:大肠杆菌,单核细胞增生乳杆菌,鼠伤寒沙门氏菌,金黄色葡萄球菌。(C和D)四种分类模型的ROC曲线。
3. 总结
本研究提出了一种简单、经济、高灵敏度的无标记SERS分析方法,用于快速分类和预测食源性致病菌。该方法基于疏水SERS底物、数据预处理技术和机器学习算法。研究人员通过优化Au@AgNPs浓度和增强底物疏水性来诱导局部浓度效应,从而提升SERS性能。结果显示,优化后的SERS底物强度比利用咖啡环效应的底物高33倍。为了提高SERS光谱的清晰度,研究人员采用了基线校正、平滑和归一化三个预处理步骤,并结合k-NN、SVM-poly、SVM-rbf和1D-CNN四种机器学习分类模型进行分析。数据预处理后,几乎所有模型的分类准确率都达到了100%。然而,本研究存在一个局限性:缺乏在实际低浓度环境中的验证,目前仅依赖于测试集进行评估。未来研究将纳入更多不同来源和环境的外部数据集,以减少选择偏差,更全面地评估模型性能。
论文链接:https://doi.org/10.1016/j.snb.2024.136963
来源:微生物安全与健康网,作者~占英。





